cGAS是宿主识别DNA并介导抗病毒天然免疫与自身免疫的关键受体。研究表明cGAS的功能与稳态受到多种翻译后修饰包括泛素化、SUMO化、磷酸化、谷氨酰化、乙酰化等的实时动态调控[1]。另外,最近有文章报道cGAS能够通过PARylation和棕榈酰化修饰负调控其介导的抗病毒免疫应答[2, 3]。然而,cGAS是否存在更多其他种类的翻译后修饰以及该修饰如何调控cGAS的活性与稳态,进而调控宿主抗病毒免疫与自身免疫等问题还有待深入研究。
ISG15 (interferon-stimulating gene 15) 是由干扰素或病原微生物诱导产生的一种类泛素分子,在宿主抵御病毒感染过程中发挥重要作用。ISG15不仅能够以细胞因子形式发挥功能,还可以通过一系列酶促反应共价结合到底物蛋白上,这一过程称为ISG15修饰。非靶向质谱的研究结果表明发生ISG15修饰的潜在底物超过300个[4, 5],然而到目前为止,cGAS是否发生ISG15修饰以及与该过程相关的生理病理学意义仍未见报道。
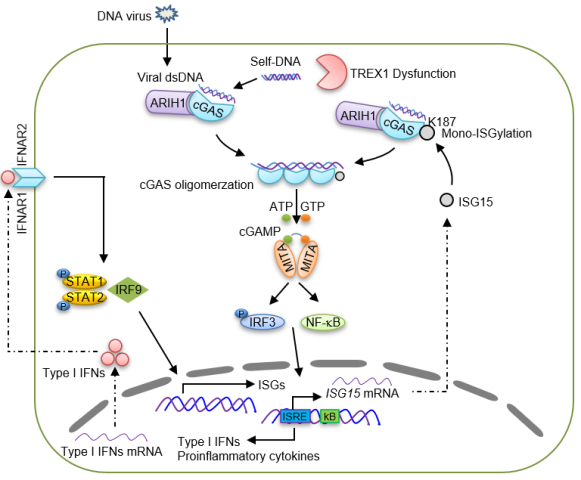
2022年10月10日,武汉大学人民医院林丹丹课题组在Nature Communications杂志在线发表了题目为 The E3 ubiquitin ligase ARIH1 promotes antiviral immunity and autoimmunity by inducing mono-ISGylation and oligomerization of cGAS 的研究论文。该团队发现E3泛素连接酶ARIH1在病毒感染后通过催化cGAS第187位赖氨酸发生单个ISG15修饰增强cGAS的寡聚化水平,从而促进抗病毒天然免疫以及Trex1缺失导致的自身免疫。

首先,该团队通过酵母双杂交实验系统地筛选了与cGAS相互作用的E3泛素连接酶,发现ARIH1与cGAS持续相互作用。在人的单核细胞系中敲低或在小鼠原代细胞中敲除ARIH1显著抑制HSV-1感染或DNA转染诱导的I型干扰素表达,髓系细胞特异敲除ARIH1的小鼠对HSV-1更加易感。
随后,该团队探究了ARIH1在Trex1敲除导致的自身免疫小鼠模型中的功能。结果表明在Trex1-/-小鼠的髓系细胞中敲除ARIH1能够挽救Trex1缺陷导致的致死性自身免疫表型,抑制脾脏生发中心B细胞的分化和CD8+ T细胞的活化,降低肝脏、肺、心脏中免疫细胞的浸润以及外周血中炎症因子和总IgG的水平。
进一步机制研究结果表明cGAS第187位赖氨酸是其寡聚化的负调控位点,ARIH1通过催化cGAS该位点发生mono-ISG15修饰,解除该位点的抑制作用,促进cGAS寡聚化和下游细胞因子产生,从而正调控cGAS介导的抗病毒天然免疫和自身免疫。
该研究阐明了ARIH1作为一个新的介导ISG15修饰的E3连接酶,通过诱导cGAS的新型翻译后修饰(mono-ISG15修饰),调控胞质内DNA诱导的抗病毒免疫与自身免疫的分子机制,暗示ARIH1可作为某些感染性疾病与自身免疫疾病的潜在分子靶标。
据悉,重庆国际免疫研究院/武汉大学医学研究院联合培养博士后熊天晨、武汉大学医学研究院博士研究生魏铭聪、硕士研究生李方旭为本文的共同第一作者,武汉大学人民医院林丹丹副教授、武汉大学中南医院张志东博士和钟波教授为本研究的共同通讯作者。
(来源:BioArt)
参考文献:
1. Zhang Z-D & Zhong B (2022) Regulation and function of the cGAS-MITA/STING axis in health and disease. Cell Insight 1(1).
2. Wang F, et al. (2022) Cytoplasmic PARP1 links the genome instability to the inhibition of antiviral immunity through PARylating cGAS. Mol Cell 82(11):2032-2049 e2037.
3. Shi C, et al. (2022) ZDHHC18 negatively regulates cGAS-mediated innate immunity through palmitoylation. Embo J 41(11):e109272.
4. Perng YC & Lenschow DJ (2018) ISG15 in antiviral immunity and beyond. Nat Rev Microbiol 16(7):423-439.
5. Zhang M, et al. (2021) ISGylation in Innate Antiviral Immunity and Pathogen Defense Responses: A Review. Front Cell Dev Biol 9:788410.